Article Outline
RでGitHub03 (Coronavirus)
公開データの場所がグーグルスプレッドシートからGitHubに移動したのでRコードを書き直しました。
新型コロナウイルスの感染状況
米ジョンズ・ホプキンス大学の新型コロナウイルスの感染状況をまとめたWebサイト
Coronavirus 2019-nCoV Global Cases by Johns Hopkins CSSE
データはGitHubから入手できます。
2019 Novel Coronavirus COVID-19 (2019-nCoV) Data Repository by Johns Hopkins CSSE
過去のデータ(archived_data)は
COVID-19 : archived_data
新型コロナウイルス関連の主なサイト
DXY.cn. Pneumonia. 2020
BNO News
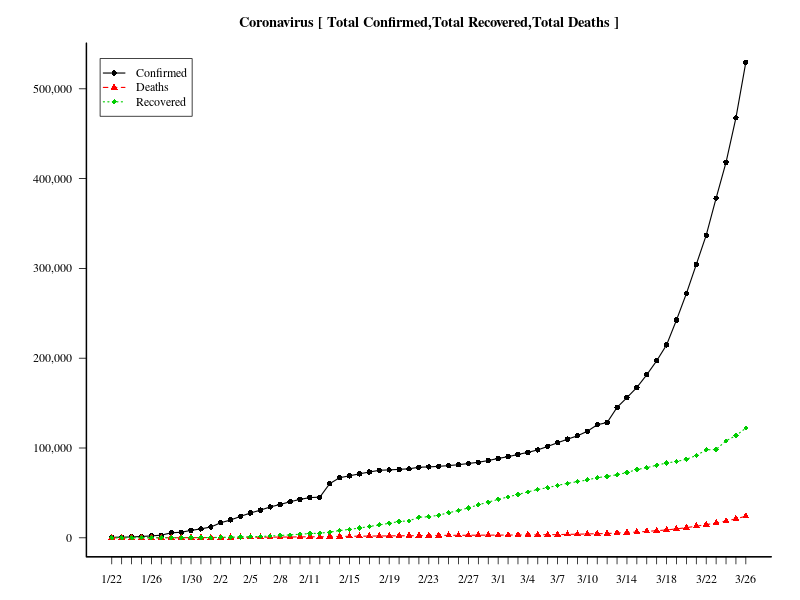
新型コロナウイルスに感染された方、回復された方、亡くなった方の数の推移(日別)
新型コロナウイルスの国、場所別感染者の推移
感染者数 10000人以上(Mainland China)
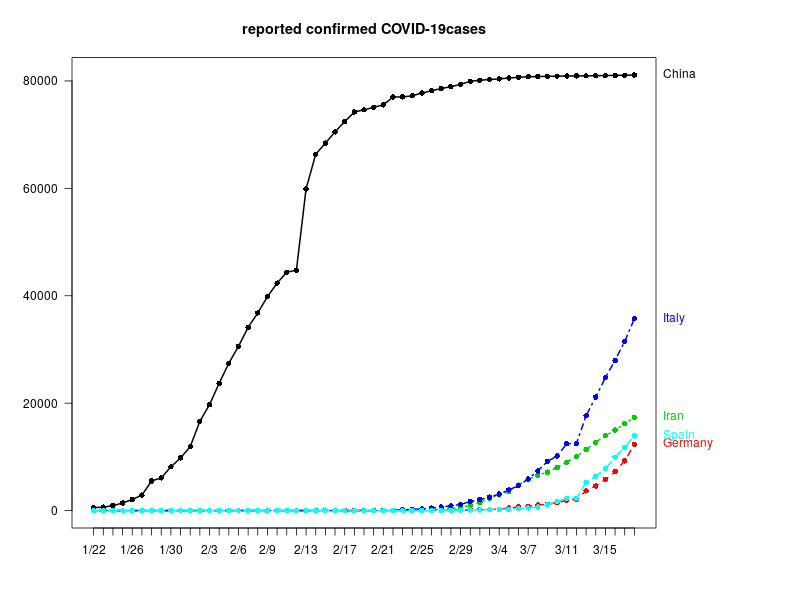
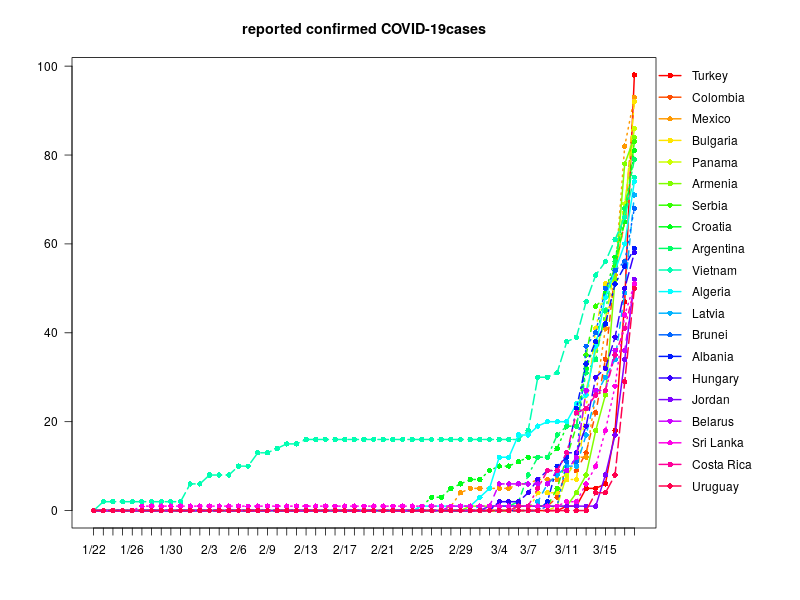
感染者数 1000人以上10000人未満
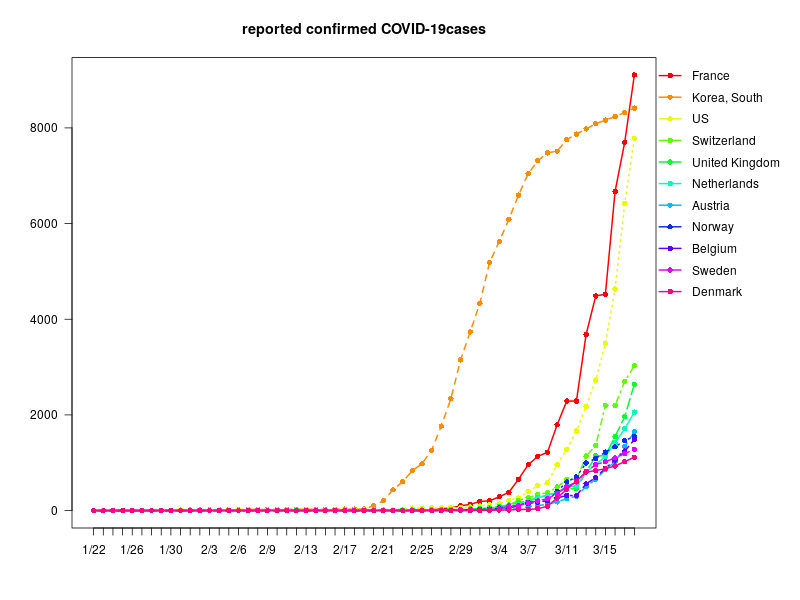
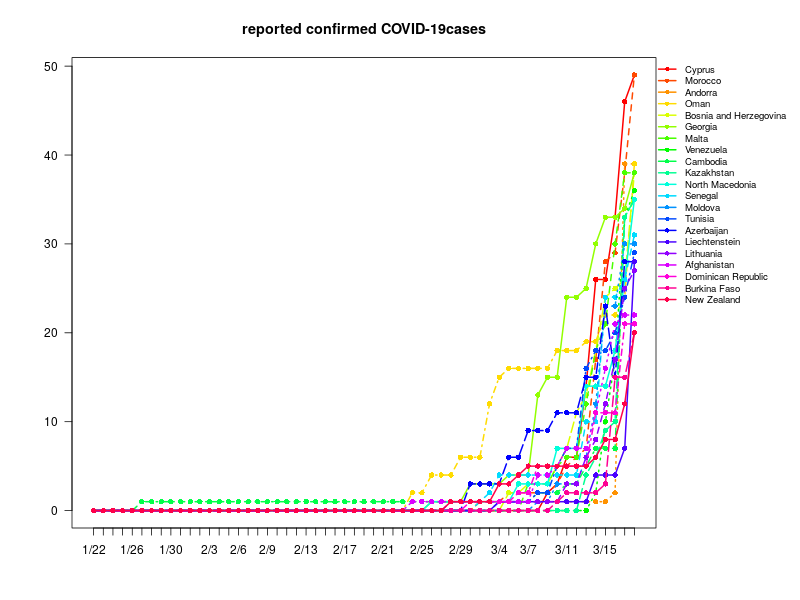
感染者数 100人以上1000人未満(OthersはDiamond Princessのこと)
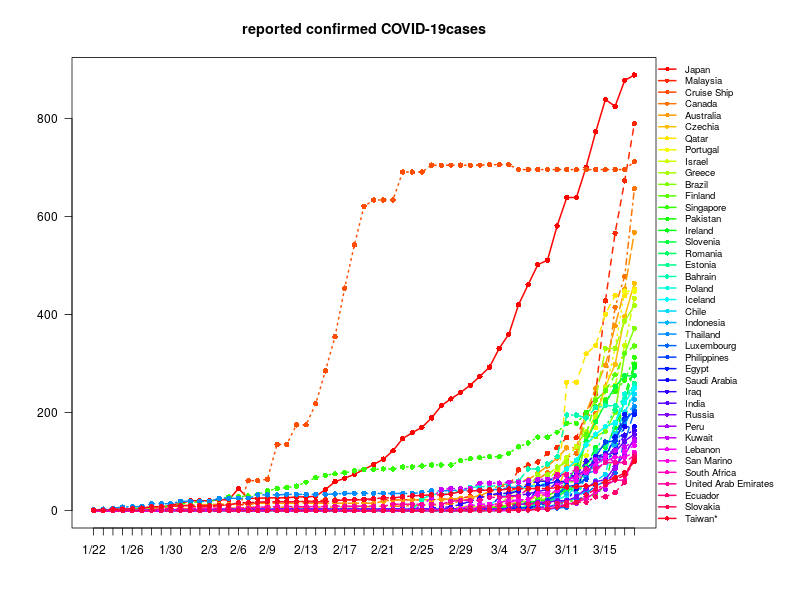
Diamond Princessや韓国、イタリアの伸び率をみると、日本はそもそも検査している数が少ないのではないかと思われる。
日本のPCR検査実施人数 報道発表資料 2020年2月より
韓国の検査数はKCDC「News Room」「Press Release」
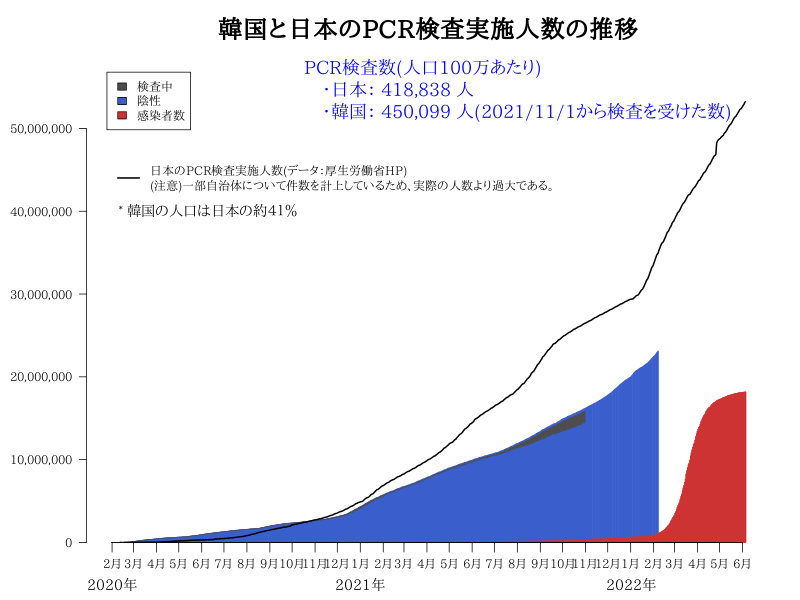
(参考)Coronavirus Disease (COVID-19) – Research and Statistics
ネット上で見つけたグラフをRで作成しました。
Alessandro Strumia(physicist) Twitter:
報告された感染者が80人を超えた時点を0とした図
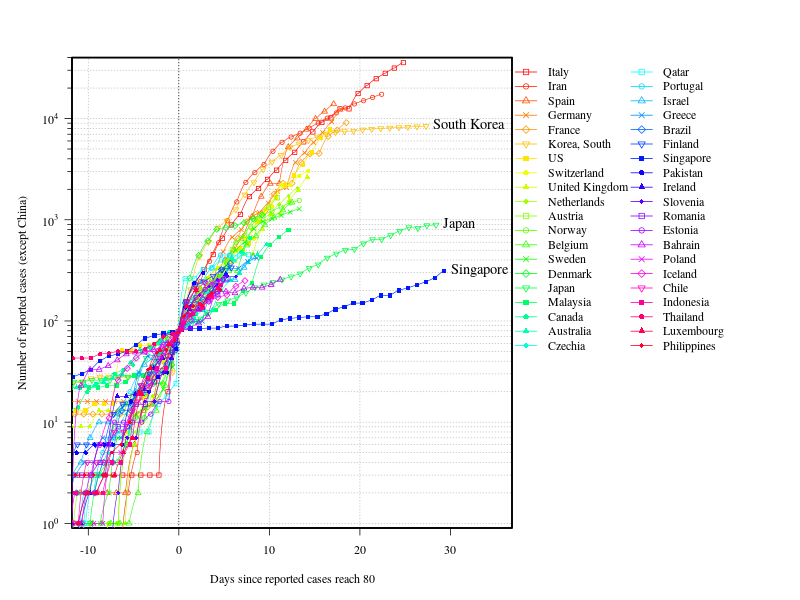
シンガポールは、報告された感染者の増加が日本より緩やかですが、
- 面積:約720平方キロメートル(東京23区と同程度)
- 人口:約564万人(うちシンガポール人・永住者は399万人)(2019年1月) 外務省HP:シンガポール共和国(Republic of Singapore)基礎データ
日本の報告された感染者の増加が他の国に比べて緩やかなのがわかります。
感染者数 50人以上100人未満
感染者数 20人以上50人未満
感染者数 10人以上20人未満
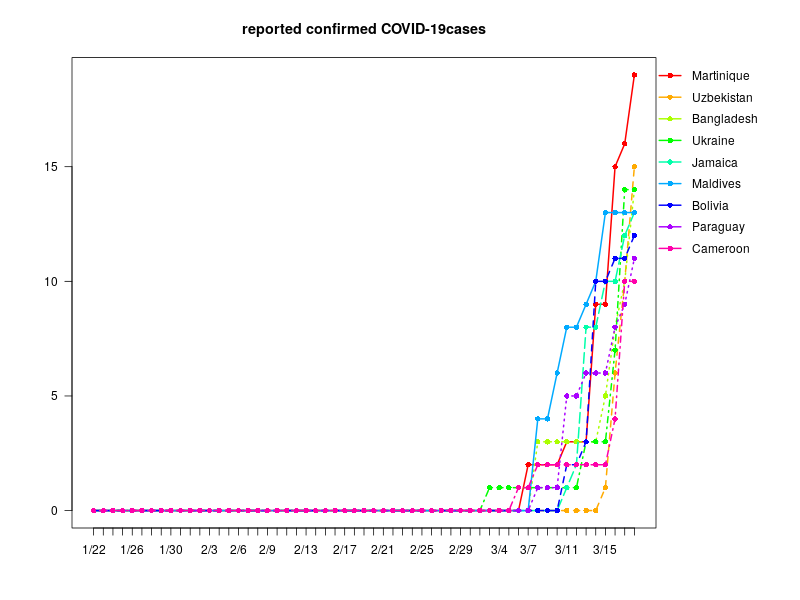
感染者数 10人未満
Rコード
パッケージの読み込み。データをGitHubから入手。(read.csvの際には、check.names=Fをつける)
# read.csvの際には、check.names=Fをつける
url<- "https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_confirmed_global.csv"
Confirmed<- read.csv(url,check.names=F)
url<- "https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_deaths_global.csv"
Deaths<- read.csv(url,check.names=F)
url<- "https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_recovered_global.csv"
Recovered<- read.csv(url,check.names=F)
#save(Confirmed,file="Confirmed.Rdata") ; save(Recovered,file="Recovered.Rdata") ; save(Deaths,file="Deaths.Rdata") time_seriesデータは一日に数回更新だったのが、日次データになっていました。
ちゃんと日順になったデータですのでコードをシンプルにしました。
# Confirmed , Deaths, Recoveredのデータ数が違うことがある。
# rbindを使うと不具合あり。もしくはエラーとなる。
nCoV1<-colSums(Confirmed[,5:ncol(Confirmed)])
nCoV2<-colSums(Deaths[,5:ncol(Deaths)])
nCoV3<-colSums(Recovered[,5:ncol(Recovered)])
lenmax<- max(c(length(nCoV1),length(nCoV2),length(nCoV3)))
nCoV<-matrix(NA,nrow=3,ncol=lenmax)
nCoV[1,1:length(nCoV1)]<- nCoV1
nCoV[2,1:length(nCoV2)]<- nCoV2
nCoV[3,1:length(nCoV3)]<- nCoV3
rownames(nCoV)<- c("Confirmed","Deaths","Recovered")
colnames(nCoV)<- names(nCoV1)感染者、回復された方、亡くなった方の数の推移(日別)
# 指数表示を抑制
options(scipen=2)
#png("Coronavirus01.png",width=800,height=600)
par(mar=c(3,6,3,2),family="serif")
matplot(t(nCoV),type="o",col=1:3,lwd=1.5,lty=1:3,pch=16:18,las=1,xaxt="n",yaxt="n",ylab="",bty="l")
box(bty="l",lwd=2)
axis(1,at=1:ncol(nCoV), labels =sub("/20","",colnames(nCoV)))
# Add comma separator to axis labels
axis(side=2, at=axTicks(2), labels=formatC(axTicks(2), format="d", big.mark=','),las=1)
legend("topleft", legend = rownames(nCoV),col=1:3,lwd=1.5,lty=1:3,pch=16:18,inset =c(0.02,0.03))
title("Coronavirus [ Total Confirmed,Total Recovered,Total Deaths ]")
#dev.off()新型コロナウイルスの国、場所別感染者の推移
# Country/Regionごとに集計
timeline<- aggregate(Confirmed[,5:ncol(Confirmed)], sum, by=list(Confirmed$"Country/Region"))
rownames(timeline)<-timeline[,1]
timeline<- timeline[,-1]
#
# データの最大値でグループ分け
# 感染者数 10000人以上(Mainland China)
G1<- timeline[apply(timeline,1,max,na.rm=T)>=10000,]
# 感染者数 1000人以上10000人未満
G2<- timeline[apply(timeline,1,max,na.rm=T)>= 1000 & apply(timeline,1,max,na.rm=T)<10000,]
# 感染者数 100人以上1000人未満
G2_2<- timeline[apply(timeline,1,max,na.rm=T)>= 100 & apply(timeline,1,max,na.rm=T)<1000,]
# 感染者数 50人以上100人未満
G3<- timeline[apply(timeline,1,max,na.rm=T)>= 50 & apply(timeline,1,max,na.rm=T)<100,]
# 感染者数 20人以上50人未満
G4<- timeline[apply(timeline,1,max,na.rm=T)>= 20 & apply(timeline,1,max,na.rm=T)<50,]
# 感染者数 10人以上20人未満
G5<- timeline[apply(timeline,1,max,na.rm=T)>= 10 & apply(timeline,1,max,na.rm=T)<20,]
# 感染者数 10人未満
G6<- timeline[apply(timeline,1,max,na.rm=T)<10,]
#
# plot
# G1
#png("CoronavirusG1.png",width=800,height=600)
par(mar=c(5,5,4,10))
plot(t(G1),type="o",pch=16,lwd=2,las=1,xlab="",ylab="",xaxt="n")
axis(1,at=1:ncol(G1),labels=sub("/20","",colnames(G1)))
text(x=par("usr")[2],y=G1[,ncol(G1)],labels=rownames(G1),pos=4,xpd=T)
title("reported confirmed COVID-19cases (mainland China)")
#dev.off()
# G2
# 降順に並べ替え
G2<-G2[order(apply(G2,1,max,na.rm=T),decreasing=T),]
col<- rainbow(nrow(G2))
#png("CoronavirusG2.png",width=800,height=600)
par(mar=c(5,5,4,10))
matplot(t(G2),type="o",pch=16,lwd=2,las=1,xlab="",ylab="",xaxt="n",col=col)
axis(1,at=1:ncol(G2),labels=sub("/20","",colnames(G2)))
# text:位置調整
#text(x=par("usr")[2],y=G2[,ncol(G2)],labels=rownames(G2),pos=4,xpd=T,col=col)
legend(x=par("usr")[2],y=par("usr")[4],legend=rownames(G2),pch=16,lwd=2,col=col,xpd=T,bty="n",y.intersp = 1.5)
title("reported confirmed COVID-19cases")
#dev.off()
# G2_2
# 降順に並べ替え
G2_2<-G2_2[order(apply(G2_2,1,max,na.rm=T),decreasing=T),]
col<- rainbow(nrow(G2_2))
#png("CoronavirusG2_2.png",width=800,height=600)
par(mar=c(5,5,4,10))
matplot(t(G2_2),type="o",pch=16,lwd=2,las=1,xlab="",ylab="",xaxt="n",col=col)
axis(1,at=1:ncol(G2_2),labels=sub("/20","",colnames(G2_2)))
# text:位置調整
#text(x=par("usr")[2],y=G2_2[,ncol(G2_2)]+c(0,0,0,30,0,-10),labels=rownames(G2_2),pos=4,xpd=T,col=col)
legend(x=par("usr")[2],y=par("usr")[4],legend=rownames(G2_2),pch=16,lwd=2,col=col,xpd=T,bty="n",y.intersp = 1.5)
title("reported confirmed COVID-19cases")
#dev.off()
# G3
# 降順に並べ替え
G3<-G3[order(apply(G3,1,max,na.rm=T),decreasing=T),]
col<- rainbow(nrow(G3))
#png("CoronavirusG3.png",width=800,height=600)
par(mar=c(5,5,4,10))
matplot(t(G3),type="o",pch=16,lwd=2,las=1,xlab="",ylab="",xaxt="n",col=col)
axis(1,at=1:ncol(G3),labels=sub("/20","",colnames(G3)))
#text(x=par("usr")[2],y=G3[,ncol(G3)],labels=rownames(G3),pos=4,xpd=T,col=col)
legend(x=par("usr")[2],y=par("usr")[4],legend=rownames(G3),pch=16,lwd=2,col=col,xpd=T,bty="n",y.intersp = 1.5)
title("reported confirmed COVID-19cases")
#dev.off()
# G4
# 降順に並べ替え
G4<-G4[order(apply(G4,1,max,na.rm=T),decreasing=T),]
col<- rainbow(nrow(G4))
#png("CoronavirusG4.png",width=800,height=600)
par(mar=c(5,5,4,10))
matplot(t(G4),type="o",pch=16,lwd=2,las=1,xlab="",ylab="",xaxt="n",col=col)
axis(1,at=1:ncol(G4),labels=sub("/20","",colnames(G4)))
# text:位置調整
#text(x=par("usr")[2],y=G4[,ncol(G4)],labels=rownames(G4),pos=4,xpd=T,col=col)
legend(x=par("usr")[2],y=par("usr")[4],legend=rownames(G4),pch=16,lwd=2,col=col,xpd=T,bty="n",y.intersp = 1.5)
title("reported confirmed COVID-19cases")
#dev.off()
# G5
# 降順に並べ替え
G5<-G5[order(apply(G5,1,max,na.rm=T),decreasing=T),]
col<- rainbow(nrow(G5))
#png("CoronavirusG5.png",width=800,height=600)
par(mar=c(5,5,4,10))
matplot(t(G5),type="o",pch=16,lwd=2,las=1,xlab="",ylab="",xaxt="n",col=col)
axis(1,at=1:ncol(G5),labels=sub("/20","",colnames(G5)))
# text:位置調整
#text(x=par("usr")[2],y=G5[,ncol(G5)]+c(0.25,-0.25,0.25,-0.25,0,0),labels=rownames(G5),pos=4,xpd=T,col=col)
legend(x=par("usr")[2],y=par("usr")[4],legend=rownames(G5),pch=16,lwd=2,col=col,xpd=T,bty="n",y.intersp = 1.5)
title("reported confirmed COVID-19cases")
#dev.off()
# G6
# 降順に並べ替え
G6<-G6[order(apply(G6,1,max,na.rm=T),decreasing=T),]
col<- rainbow(nrow(G6))
#png("CoronavirusG6.png",width=800,height=600)
par(mar=c(5,5,4,15))
matplot(t(G6),type="o",pch=16,lwd=2,las=1,xlab="",ylab="",xaxt="n",col=col)
axis(1,at=1:ncol(G6),labels=sub("/20","",colnames(G6)))
#legend(x=par("usr")[2],y=par("usr")[4],legend=rownames(G6),pch=16,lwd=2,col=col,xpd=T,bty="n",y.intersp = 1,cex=0.8,ncol=2)
legend(x="topleft",legend=rownames(G6),pch=16,lwd=2,col=col,xpd=T,bty="n",y.intersp = 1,cex=0.9,ncol=2)
title("reported confirmed COVID-19cases")
#dev.off()報告された感染者が80人を超えた時点を0とした図
# Country/Regionごとに集計
timeline<- aggregate(Confirmed[,5:ncol(Confirmed)], sum, by=list(Confirmed$"Country/Region"))
rownames(timeline)<-timeline[,1]
timeline<- timeline[,-1]
#
# 感染者数 200人以上20000人未満
min<- 200
max<- 20000
G1_2<- timeline[apply(timeline,1,max,na.rm=T)>= min & apply(timeline,1,max,na.rm=T)< max,]
# Cruise Shipを除く
G1_2<-G1_2[grep("Cruise Ship",rownames(G1_2),invert =T),]
G1_2<-G1_2[order(apply(G1_2,1,max,na.rm=T),decreasing=T),]
col<- rainbow(nrow(G1_2))
#
#Starting point
Sp<- 80 # Starting pointとする感染者数
length<- 10 # Starting pointとする感染者数に合わせるための調整
xlim=c(-22,30) # 範囲
col<- rainbow(nrow(G1_2))
pch<- rep(c(0,1,2,4,5,6,15,16,17,18),5)
#png("CoronavirusG1_2L.png",width=800,height=600)
par(mar=c(5,5,4,2),family="serif")
#
plot(1,1,type="n",xlab=paste0("Days since reported cases reach ",Sp),
ylab="Number of reported cases (except China)",yaxt="n",log="y",
ylim=c(0.9,20000),xlim=xlim,yaxs="i")
abline(v=seq(-20,20,10),col=c("gray","gray","black","gray","gray"),lty=3)
for(i in 0:5){
abline(h=seq(1,9)*10^i,col="gray",lty=3)
}
box(lwd=2.5)
#
for (i in 1:length(col)){
p0<- as.numeric(G1_2[i,])
p<-NULL
for (j in 1:(length(p0)-1)){
p<- c(p,seq(p0[j],p0[j+1],length=length))
}
p1<- length(p[p<Sp])
p2<- length(p[p>=Sp])
lines(seq(-p1/length,(p2/length - 1/length),1/length),p,lwd=0.8,col=col[i])
points(seq(-p1,p2,length)/length,p0,cex=0.8,col=col[i],pch=pch[i])
if (i== grep("Japan",rownames(G1_2))){
text(x=p2[length(p2)]/length,y=p0[length(p0)],labels="Japan",cex=1.2,col="black",pos=4)
}
if (i== grep("Korea, South",rownames(G1_2))){
text(x=p2[length(p2)]/length,y=p0[length(p0)],labels="South Korea",cex=1.2,col="black",pos=4)
}
if (i== grep("Singapore",rownames(G1_2))){
text(x=p2[length(p2)]/length,y=p0[length(p0)],labels="Singapore",cex=1.2,col="black",pos=4)
}
}
for(i in 0:5){
axis(side=2, at=10^i, labels=bquote(10^.(i)) ,las=1)
axis(side=2, at=seq(2,9)*10^i, tck=-0.01,labels=F)
}
legend(x="topleft",inset=c(0.03,0.01),ncol=2,legend=rownames(G1_2),pch=pch,lwd=1,col=col,xpd=T,
bty="n",x.intersp= 1,y.intersp =1.1,cex=1.2)
#legend(x=par("usr")[2],y=10^par("usr")[4],legend=rownames(G1_2),pch=pch,lwd=1,col=col,xpd=T,bty="n",cex=1)
# dev.off()現在は使っていないRコードです。lubridateパッケージの覚書として残しています。
library(xts)
library(lubridate)
#
# read.csvの際には、check.names=Fをつける
url<- "https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_19-covid-Confirmed.csv"
Confirmed<- read.csv(url,check.names=F)
url<- "https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_19-covid-Recovered.csv"
Recovered<- read.csv(url,check.names=F)
url<- "https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_19-covid-Deaths.csv"
Deaths<- read.csv(url,check.names=F)time_seriesデータは一日に数回更新だったのが、日次データになっていました。
#lubridateパッケージを使うのが便利
t<-mdy(colnames(Confirmed)[5:ncol(Confirmed)])
nCoV<-data.frame(date=t,Confirmed=as.vector(colSums(Confirmed[,5:ncol(Confirmed)],na.rm=T)))
#
t<-mdy(colnames(Recovered)[5:ncol(Recovered)])
d<-data.frame(date=t,Recovered=as.vector(colSums(Recovered[,5:ncol(Recovered)],na.rm=T)))
# merge
nCoV<-merge(nCoV,d,by="date")
#
t<-mdy(colnames(Deaths)[5:ncol(Deaths)])
d<-data.frame(date=t,Deaths=as.vector(colSums(Deaths[,5:ncol(Deaths)],na.rm=T)))
nCoV<-merge(nCoV,d,by="date")感染者、回復された方、亡くなった方の数の推移(日別)
# png("Coronavirus01.png",width=800,height=600)
par(mar=c(3,5,3,2))
matplot(nCoV[,2:4],type="o",col=1:3,lwd=1.5,lty=1:3,pch=16:18,las=1,xaxt="n",ylab="")
axis(1,at=1:nrow(nCoV), labels =sub("2020-","",nCoV[,1] ))
legend("topleft", legend = colnames(nCoV[,2:4]),col=1:3,lwd=1.5,lty=1:3,pch=16:18,inset =c(0.02,0.03))
title("Coronavirus [ Total Confirmed,Total Recovered,Total Deaths ]")
# dev.off()