Rで塗り分け地図(コロプレス図)(新型コロナウイルス)
(参考:コロプレス図作成)5. Plotting Simple Features
使用するデータ(厚生労働省:pdfファイル)
各都道府県の検査陽性者の状況(空港検疫、チャーター便案件を除く国内事例)
「PCR検査実施人数は、一部自治体について件数を計上している。」ので県別に比較はできない。 ここでは"都道府県名","PCR検査陽性者","死亡"をとりあげます。 都道府県の人口は地図データに入っているものを使います。
pdfファイルから取り出して、加工した表
(注意)「PCR検査実施人数は、一部自治体について件数を計上している。」ので県別に比較はできない。
| 都道府県名 | 陽性者数 | 検査実施人数 | 入院治療等 | うち重症 | 退院又は療養解除 | 死亡者数 | 確認中 |
|---|---|---|---|---|---|---|---|
| 北海道 | 1413 | 28554 | 73 | 3 | 1237 | 103 | 0 |
| 青森 | 32 | 1445 | 2 | 0 | 29 | 1 | 0 |
| 岩手 | 3 | 1480 | 3 | 0 | 0 | 0 | 0 |
| 宮城 | 158 | 5843 | 17 | 0 | 140 | 1 | 0 |
| 秋田 | 18 | 1146 | 2 | 0 | 16 | 0 | 0 |
| 山形 | 76 | 2894 | 1 | 0 | 75 | 1 | 1 |
| 福島 | 89 | 9845 | 6 | 0 | 83 | 0 | 0 |
| 茨城 | 280 | 7081 | 54 | 1 | 216 | 10 | 0 |
| 栃木 | 195 | 13517 | 47 | 0 | 139 | 0 | 9 |
| 群馬 | 190 | 8173 | 30 | 1 | 141 | 19 | 0 |
| 埼玉 | 2313 | 66219 | 504 | 2 | 1735 | 74 | 0 |
| 千葉 | 1656 | 28022 | 370 | 4 | 1237 | 49 | 0 |
| 東京 | 12691 | 187764 | 2744 | 16 | 9615 | 332 | 0 |
| 神奈川 | 2484 | 18126 | 341 | 10 | 2044 | 99 | 0 |
| 新潟 | 111 | 6417 | 22 | 0 | 88 | 0 | 1 |
| 富山 | 238 | 5323 | 6 | 0 | 210 | 22 | 0 |
| 石川 | 321 | 3266 | 19 | 3 | 275 | 27 | 0 |
| 福井 | 139 | 4852 | 14 | 0 | 117 | 8 | 0 |
| 山梨 | 94 | 7260 | 14 | 0 | 79 | 1 | 0 |
| 長野 | 105 | 7146 | 22 | 0 | 83 | NA | 0 |
| 岐阜 | 331 | 10353 | 123 | 0 | 201 | 7 | 0 |
| 静岡 | 269 | 13280 | 140 | 1 | 128 | 1 | 0 |
| 愛知 | 1609 | 22910 | 1015 | 2 | 553 | 35 | 6 |
| 三重 | 91 | 4322 | 35 | 0 | 53 | 1 | 2 |
| 滋賀 | 171 | 4368 | 57 | 2 | 113 | 1 | 0 |
| 京都 | 758 | 17749 | 210 | 2 | 528 | 20 | 0 |
| 大阪 | 4057 | 72661 | 1262 | 19 | 2702 | 90 | 3 |
| 兵庫 | 1158 | 25952 | 236 | 5 | 877 | 45 | 0 |
| 奈良 | 235 | 8733 | 55 | 0 | 178 | 2 | 0 |
| 和歌山 | 150 | 6159 | 43 | 2 | 102 | 3 | 2 |
| 鳥取 | 15 | 2785 | 10 | 0 | 5 | 0 | 0 |
| 島根 | 29 | 2916 | 4 | 0 | 25 | 0 | 0 |
| 岡山 | 79 | 2974 | 32 | NA | 42 | NA | 5 |
| 広島 | 312 | 11610 | 84 | 0 | 222 | 3 | 3 |
| 山口 | 53 | 3872 | 12 | 0 | 41 | 0 | 0 |
| 徳島 | 25 | 1788 | 13 | 0 | 8 | 1 | 3 |
| 香川 | 46 | 4635 | 4 | 0 | 42 | 0 | 0 |
| 愛媛 | 89 | 3006 | 2 | 0 | 82 | 5 | 0 |
| 高知 | 80 | 2252 | 4 | 0 | 73 | 3 | 0 |
| 福岡 | 1756 | 23888 | 661 | 4 | 1062 | 33 | 0 |
| 佐賀 | 82 | 2407 | 33 | 0 | 53 | 0 | 4 |
| 長崎 | 74 | 6830 | 45 | NA | 27 | 2 | 0 |
| 熊本 | 191 | 5498 | 140 | 0 | 48 | 3 | 0 |
| 大分 | 66 | 6680 | 6 | 0 | 59 | 1 | 0 |
| 宮崎 | 121 | 3985 | 100 | 0 | 21 | 0 | 0 |
| 鹿児島 | 236 | 10464 | 84 | 0 | 152 | 0 | 0 |
| 沖縄 | 395 | 8033 | 244 | 3 | 148 | 7 | 0 |
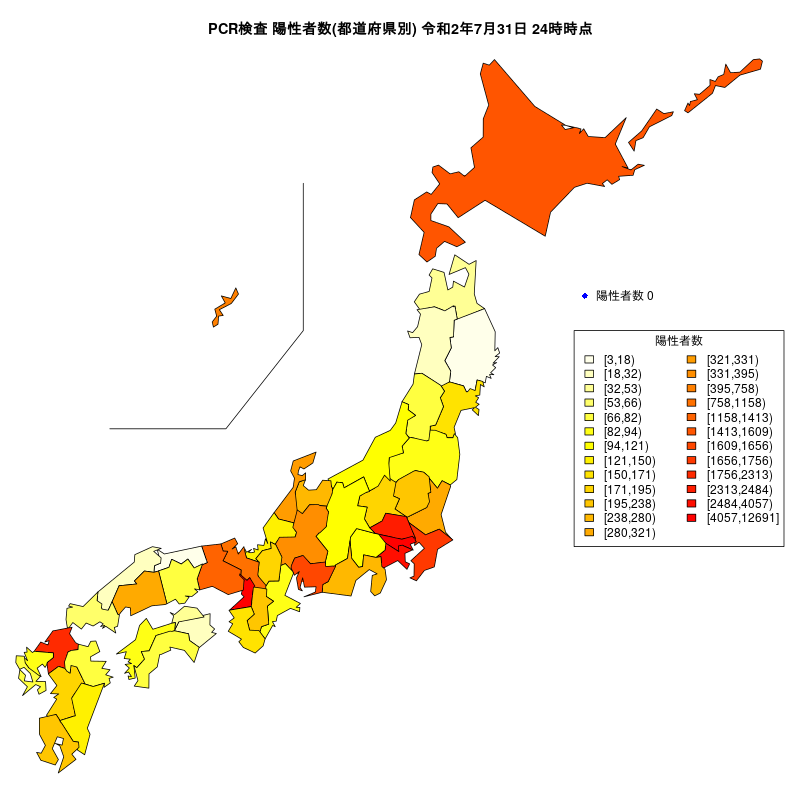
陽性者数
ここからは、人口を考慮に入れて作図します。
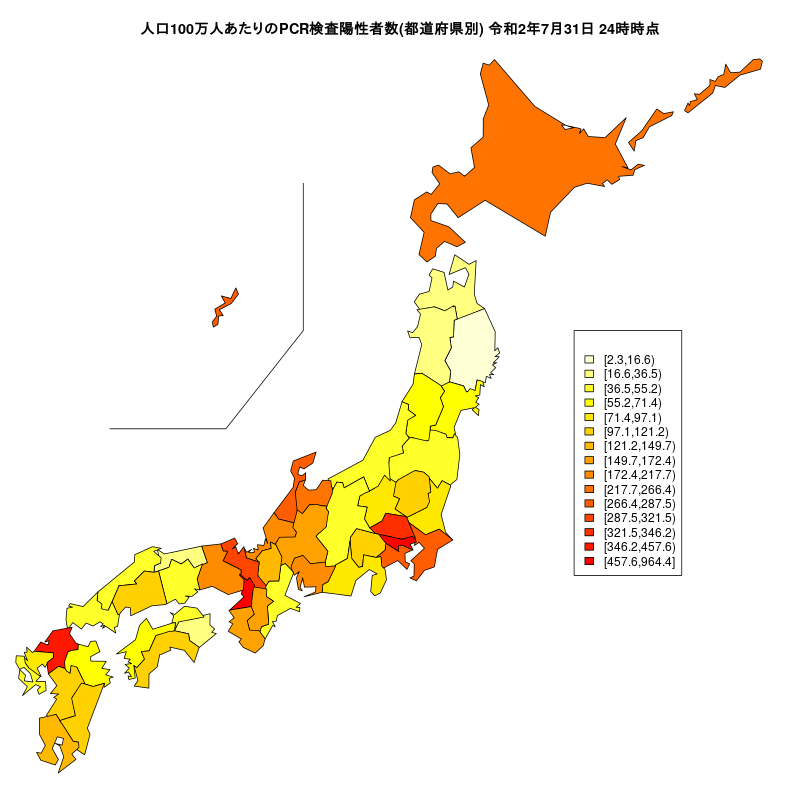
人口100万人あたりのPCR検査陽性者数
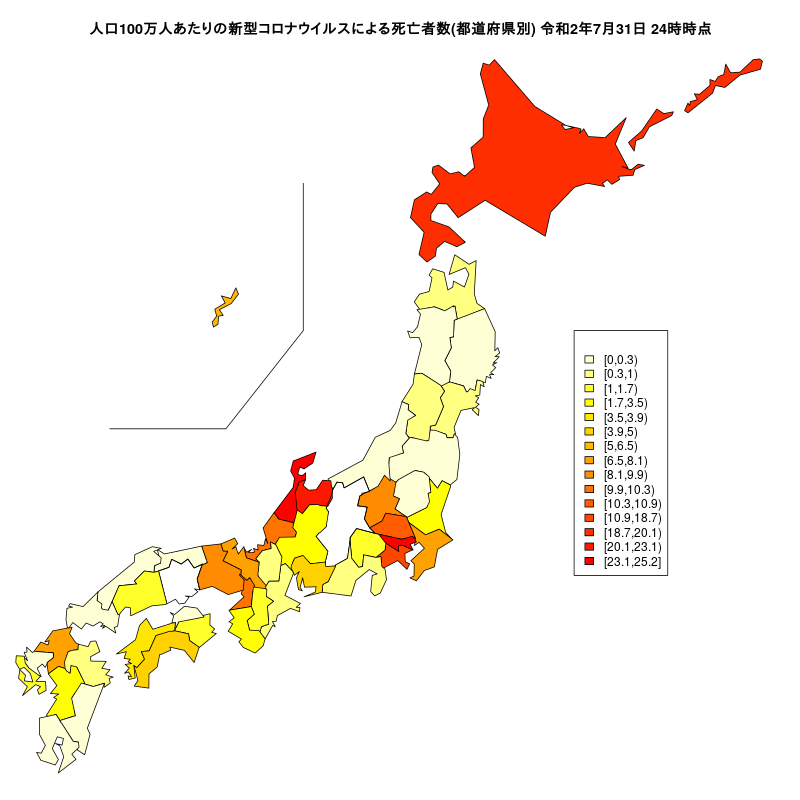
人口100万人あたりの新型コロナウイルスによる死亡者数
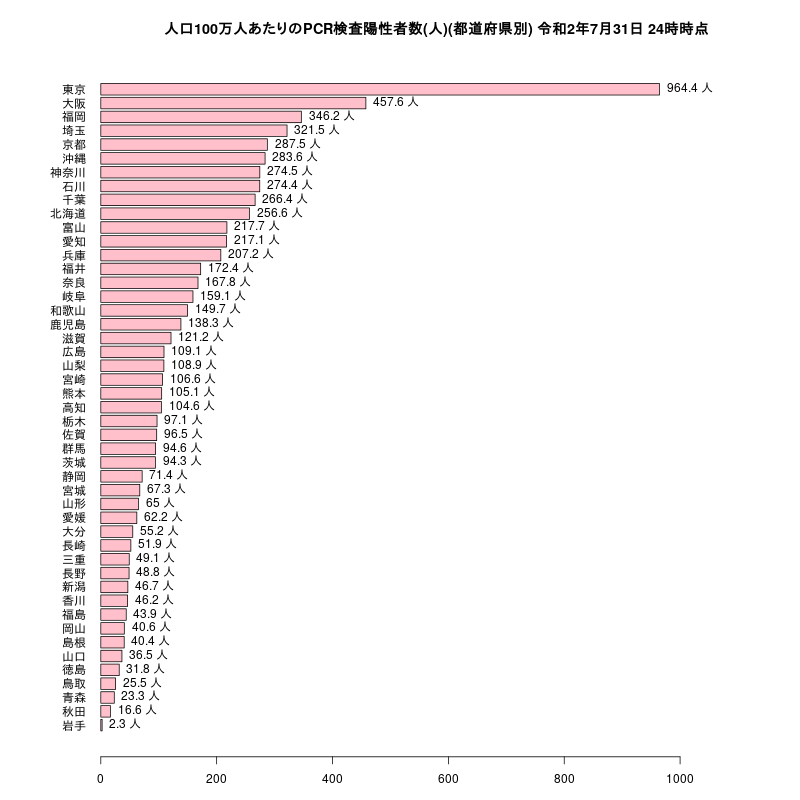
人口100万人あたりのPCR検査陽性者数の棒グラフ(都道府県別)の棒グラフ
散布図(人口100万人あたりのPCR検査陽性者数,人口100万人あたりの新型コロナウイルスによる死亡者数)
人口100万人あたりのPCR検査陽性者数80人以下、人口100万人あたりの新型コロナウイルスによる死亡者数5人以下の都道府県のみプロット
Rコード
pdfファイルからデータの取り込み。加工。
- pdfファイルからデータの取り込み(pdftools::pdf_text)
- 余分な部分を削除(sub,gsub関数)
- data.frameへ(read.table(text=txt,stringsAsFactors = F) )
# install.packages("pdftools")
library(pdftools)
library(knitr)
#
res <- pdf_text("https://www.mhlw.go.jp/content/10906000/000655357.pdf")
#res <- pdf_text("./covid19/000655357.pdf")
period<-paste0("令和",sub("時点.*$","",sub("^.*令和","",res)),"時点")
# c("都道府県名","PCR検査陽性者","PCR検査実施人数","入院治療等を要する者","うち重症","退院又は療養解除となった者の数","死亡","確認中")
txt<- gsub(",","",res)
txt<- sub("(その他).*$","",txt)
txt<- sub("^.*北海道","北海道",txt)
txt<- gsub(" +"," ",txt)
txt<- gsub(" ",",",txt)
txt<- gsub(" ","",txt)
txt<- gsub("※4","",txt)
txt<- gsub("※5","",txt)
txt<- gsub("-","NA",txt)
txt<- gsub("\n,","\n",txt)
txt<- gsub("G48の関数(=O47=★当日デー","",txt)
# stringsAsFactors = Fをつけて、Factor化を防ぐ。
Jtest<- read.csv(text=txt,stringsAsFactors = F,h=F)
colnames(Jtest)<- c("都道府県名","陽性者数","検査実施人数","入院治療等","うち重症","退院又は療養解除","死亡者数","確認中")
# 「PCR検査実施人数は、一部自治体について件数を計上している。」ので県別に比較はできない。
# ここでは"都道府県名","PCR検査陽性者","死亡"をとりあげる。
summary(Jtest)
kable(Jtest)
save(Jtest,file="Jtest.Rdata")
#load("Jtest.Rdata")塗り分け地図作成
パッケージの読み込み。都道府県の並びを確認。
library(sf)
library(NipponMap)
library(BAMMtools)
shp <- system.file("shapes/jpn.shp", package = "NipponMap")[1]
m <- sf::read_sf(shp)
m$name [1] "Hokkaido" "Aomori" "Iwate" "Miyagi" "Akita" "Yamagata"
[7] "Fukushima" "Ibaraki" "Tochigi" "Gunma" "Saitama" "Chiba"
[13] "Tokyo" "Kanagawa" "Niigata" "Toyama" "Ishikawa" "Fukui"
[19] "Yamanashi" "Nagano" "Gifu" "Shizuoka" "Aichi" "Mie"
[25] "Shiga" "Kyoto" "Osaka" "Hyogo" "Nara" "Wakayama"
[31] "Tottori" "Shimane" "Okayama" "Hiroshima" "Yamaguchi" "Tokushima"
[37] "Kagawa" "Ehime" "Kochi" "Fukuoka" "Saga" "Nagasaki"
[43] "Kumamoto" "Oita" "Miyazaki" "Kagoshima" "Okinawa"
(注意)上のJtestデータの都道府県の並びがこの順序になっているか確認する。
地図作成
BAMMtoolsパッケージのgetJenksBreaks関数を使い値の区切りを決めます。
グループの数も指定せずに済むようにしました。
陽性者数
dat<- Jtest$陽性者数
# legendのタイトル
ltitle<- "陽性者数"
# グラフのタイトル
title<- "PCR検査 陽性者数(都道府県別)"
#
##### ここ以降のRコードは共通 #####
#
# Jenksの自然分類法で分ける最大
i <- length(dat)
brk <- getJenksBreaks(dat,k=i+1)
while (length(unique(brk)) != length(brk)) {
brk <- getJenksBreaks(dat,k=i+1)
i=i-1
}
# legendのlabelを作成
labels<- as.vector(cut(brk[1:length(brk)-1],breaks=brk,include.lowest=T,right =F, dig.lab=5))
# 塗りつぶしに使うカラーパレット:rev関数で 白->赤
color<- rev(heat.colors(length(brk)-1))
cols<-as.vector(cut(dat, breaks=brk,labels =color,include.lowest=T,right =F))
#png("covmap21.png",width=800,height=800)
par(mar=c(0,0,4,0))
JapanPrefMap(col=cols)
legend(x=144,y=40, legend=labels, fill=color,title =ltitle,ncol=2)
title(paste(title,period))
# 陽性者数0の県に青丸をつける場合
shp <- system.file("shapes/jpn.shp", package = "NipponMap")[1]
m <- sf::read_sf(shp)
st_crs(m) <- "+proj=longlat +datum=WGS84"
m[,"Cases"]<- dat
m0 <- m
m0$geometry[[47]] <- m0$geometry[[47]] + c(3.5,14)
zero<- m0[m0$Cases==0,]
plot(st_geometry(st_centroid(zero)), pch =16, col ="blue", add = TRUE)
legend(x=144,y=41, legend="陽性者数 0",pch =16, col ="blue",bty="n")
#dev.off()人口100万人あたりのPCR検査陽性者数
都道府県の人口は地図データに入っているものを使います。
# 人口100万人あたりの陽性者数
dat<- round((1000000*Jtest$陽性者数)/m$population,1)
# legendのタイトル
ltitle<- ""
# グラフのタイトル
title<- "人口100万人あたりのPCR検査陽性者数(都道府県別)"
#
##### ここ以降のRコードは共通 #####
#
# Jenksの自然分類法で分ける最大
i <- length(dat)
brk <- getJenksBreaks(dat,k=i+1)
while (length(unique(brk)) != length(brk)) {
brk <- getJenksBreaks(dat,k=i+1)
i=i-1
}
# legendのlabelを作成
labels<- as.vector(cut(brk[1:length(brk)-1],breaks=brk,include.lowest=T,right =F, dig.lab=5))
# 塗りつぶしに使うカラーパレット:rev関数で 白->赤
color<- rev(heat.colors(length(brk)-1))
cols<-as.vector(cut(dat, breaks=brk,labels =color,include.lowest=T,right =F))
#png("covmap22.png",width=800,height=800)
par(mar=c(0,0,4,0))
JapanPrefMap(col=cols)
legend(x=144,y=40, legend=labels, fill=color,title =ltitle)
title(paste(title,period))
#dev.off()人口100万人あたりの新型コロナウイルスによる死亡者数
# 人口100万人あたりの死亡者数
dat<- round((1000000*Jtest$死亡者数)/m$population,1)
# legendのタイトル
ltitle<- ""
# グラフのタイトル
title<- "人口100万人あたりの新型コロナウイルスによる死亡者数(都道府県別)"
#
##### ここ以降のRコードは共通 #####
#
# Jenksの自然分類法で分ける最大
i <- length(dat)
brk <- getJenksBreaks(dat,k=i+1)
while (length(unique(brk)) != length(brk)) {
brk <- getJenksBreaks(dat,k=i+1)
i=i-1
}
# legendのlabelを作成
labels<- as.vector(cut(brk[1:length(brk)-1],breaks=brk,include.lowest=T,right =F, dig.lab=5))
# 塗りつぶしに使うカラーパレット:rev関数で 白->赤
color<- rev(heat.colors(length(brk)-1))
cols<-as.vector(cut(dat, breaks=brk,labels =color,include.lowest=T,right =F))
#png("covmap23.png",width=800,height=800)
par(mar=c(0,0,4,0))
JapanPrefMap(col=cols)
legend(x=144,y=40, legend=labels, fill=color,title =ltitle)
title(paste(title,period))
#dev.off()人口100万人あたりのPCR検査陽性者数の棒グラフ
dat<- Jtest[,c("都道府県名","陽性者数")]
dat$人口100万人あたりのPCR検査陽性者数<- round((1000000*Jtest$陽性者数)/m$population,1)
dat<- dat[order(dat$人口100万人あたりのPCR検査陽性者数),]
# グラフのタイトル
title<- "人口100万人あたりのPCR検査陽性者数(人)(都道府県別)"
#
#png("covidB21.png",width=800,height=800)
par(mar=c(3,7,4,2))
b<- barplot(dat$人口100万人あたりのPCR検査陽性者数,names=dat$都道府県名,horiz=T,col="pink",las=1,
xlim=c(0,max(dat$人口100万人あたりのPCR検査陽性者数)*1.2))
text(x=dat$人口100万人あたりのPCR検査陽性者数,y=b,labels=paste(dat$人口100万人あたりのPCR検査陽性者数,"人"),pos=4)
title(paste(title,period))
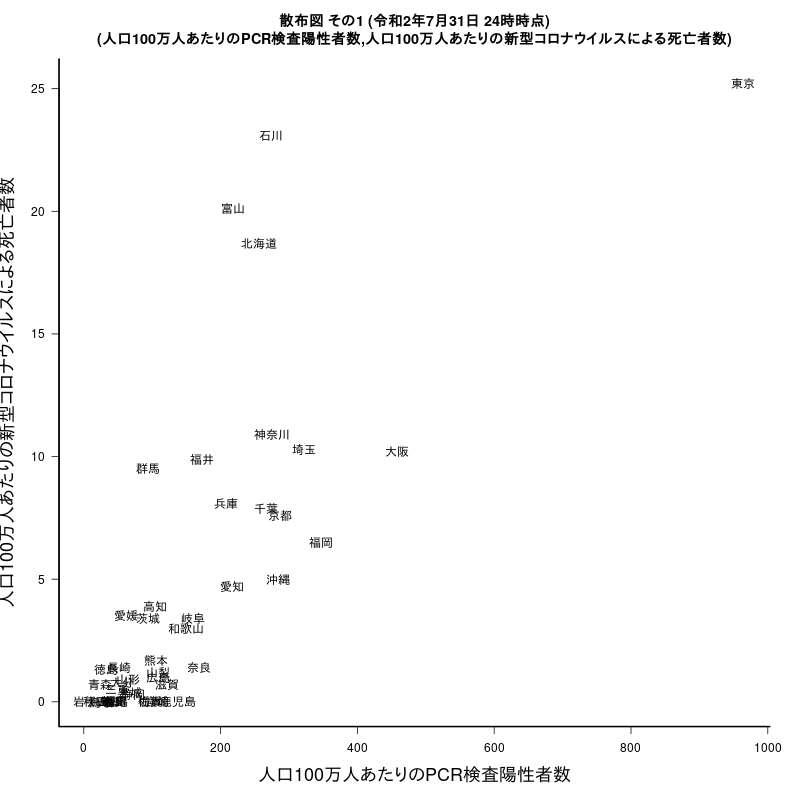
#dev.off()散布図(人口100万人あたりのPCR検査陽性者数,人口100万人あたりの新型コロナウイルスによる死亡者数)
x<- round((1000000*Jtest$陽性者数)/m$population,1)
y<- round((1000000*Jtest$死亡者数)/m$population,1)
#png("covidP21.png",width=800,height=800)
plot(x,y,type="n",bty="l",las=1,
xlab="人口100万人あたりのPCR検査陽性者数",ylab="人口100万人あたりの新型コロナウイルスによる死亡者数",cex.lab=1.5)
box(bty="l",lwd=2)
text(x=x,y=y,labels=Jtest$都道府県名)
title(paste0("散布図 その1 (",period,")\n(人口100万人あたりのPCR検査陽性者数,人口100万人あたりの新型コロナウイルスによる死亡者数)"))
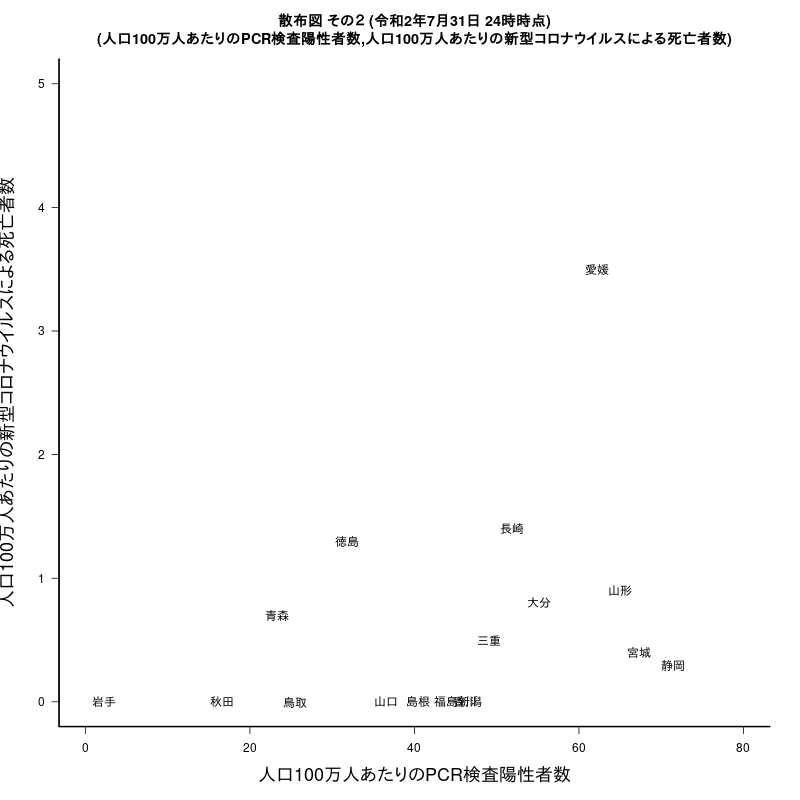
#dev.off()人口100万人あたりのPCR検査陽性者数80人以下、人口100万人あたりの新型コロナウイルスによる死亡者数5人以下の都道府県のみプロット
x<- round((1000000*Jtest$陽性者数)/m$population,1)
y<- round((1000000*Jtest$死亡者数)/m$population,1)
#png("covidP22.png",width=800,height=800)
plot(x,y,type="n",bty="l",las=1,xlim=c(0,80),ylim=c(0,5),
xlab="人口100万人あたりのPCR検査陽性者数",ylab="人口100万人あたりの新型コロナウイルスによる死亡者数",cex.lab=1.5)
box(bty="l",lwd=2)
text(x=x,y=y,labels=Jtest$都道府県名)
title(paste0("散布図 その2 (",period,")\n(人口100万人あたりのPCR検査陽性者数,人口100万人あたりの新型コロナウイルスによる死亡者数)"))
#dev.off()