Article Outline
デンドログラムのラベルを縦書きにする(新型コロナウイルス:Coronavirus)
- tategaki 関数(簡潔でスッキリとした関数)を参考にさせてもらいました。
(使用するデータ)
NHK:新型コロナ データ
時系列クラスタリングするデータ
累計データだとすべて全区間右上がりもしくは横々なので日別データを使う。
日別人口100万人あたり感染者数 [ データ:NHK ]

- 時系列クラスタリングしてみたら、外れ値に引きずられぎみなので7日移動平均を算出。
日別人口100万人あたり感染者数の7日移動平均 [ データ:NHK ]

- これを使います。
デンドログラム
デンドログラム(horiz=T)
デンドログラム : ラベルを縦書き
デンドログラム : ラベルを縦書き(dendextendパッケージを使う)
Rコード
パッケージ読み込み、データ読み込み
library(xts)
library(TTR)
library(sf)
library(NipponMap)
#
nhkC<- read.csv("https://www3.nhk.or.jp/n-data/opendata/coronavirus/nhk_news_covid19_prefectures_daily_data.csv")
#
# 都道府県別人口
shp <- system.file("shapes/jpn.shp", package = "NipponMap")[1]
m <- sf::read_sf(shp)日別人口100万人あたり感染者数 [ データ:NHK ]
code<- 1:47
column<- c(1,4)
perP<- 1000000
#
Cddata<- nhkC[nhkC$都道府県コード==code[1],column]
Cddata.xts<- as.xts(read.zoo(Cddata, format="%Y/%m/%d"))
Cddata.xts<- round(Cddata.xts[,1]*perP/m$population[1],2)
#
for (i in code[-1]){
Cddata<- nhkC[nhkC$都道府県コード== i,column]
tmp.xts<- as.xts(read.zoo(Cddata, format="%Y/%m/%d"))
tmp.xts<- round(tmp.xts[,1]*perP/m$population[i],2)
Cddata.xts<- merge(Cddata.xts,tmp.xts)
}
# NA<- 0
coredata(Cddata.xts)[is.na(Cddata.xts)]<- 0
colnames(Cddata.xts)<- unique(nhkC[nhkC$都道府県コード==code,"都道府県名"])
#
#png("Cdendro00.png",width=800,height=600)
plot(Cddata.xts)
#dev.off()日別人口100万人あたり感染者数の7日移動平均 [ データ:NHK ]
dat<- data.frame(apply(coredata(Cddata.xts),2,SMA,n=7)[-c(1:6),])
#png("Cdendro000.png",width=800,height=600)
plot(xts(read.zoo(data.frame(index(Cddata.xts)[-c(1:6)],dat))))
#dev.off()時系列クラスタリング
library(TSclust)
#
# 日別人口100万人あたり感染者数の7日移動平均
### CORT距離で距離行列を作成
d <- diss(dat, "CORT")
# hclust は method = "ward.D2"
h <- hclust(d,method="ward.D2")デンドログラム
#png("Cdendro01.png",width=800,height=600)
par(mar=c(3,5,4,2),cex=1.2)
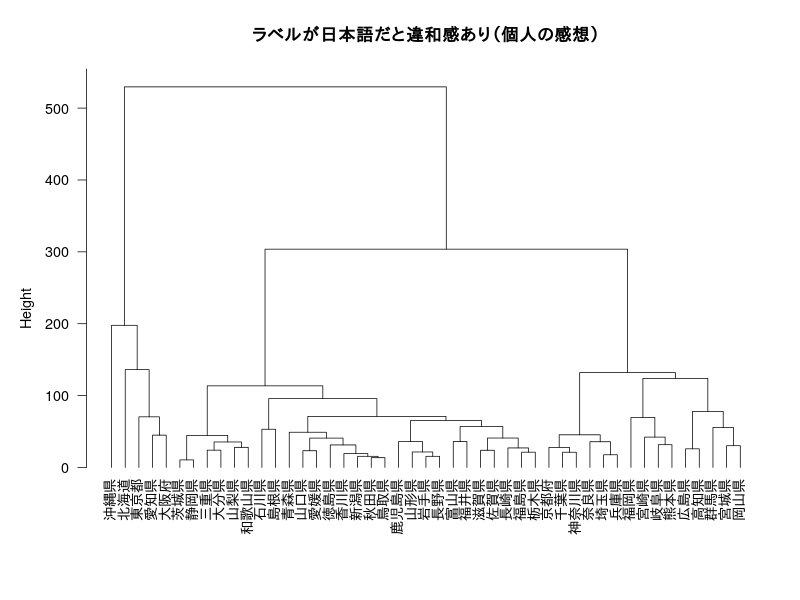
plot(h, hang=-1,las=1,main="ラベルが日本語だと違和感あり(個人の感想)")
#dev.off()デンドログラム(horiz=T)
#png("Cdendro02.png",width=800,height=800)
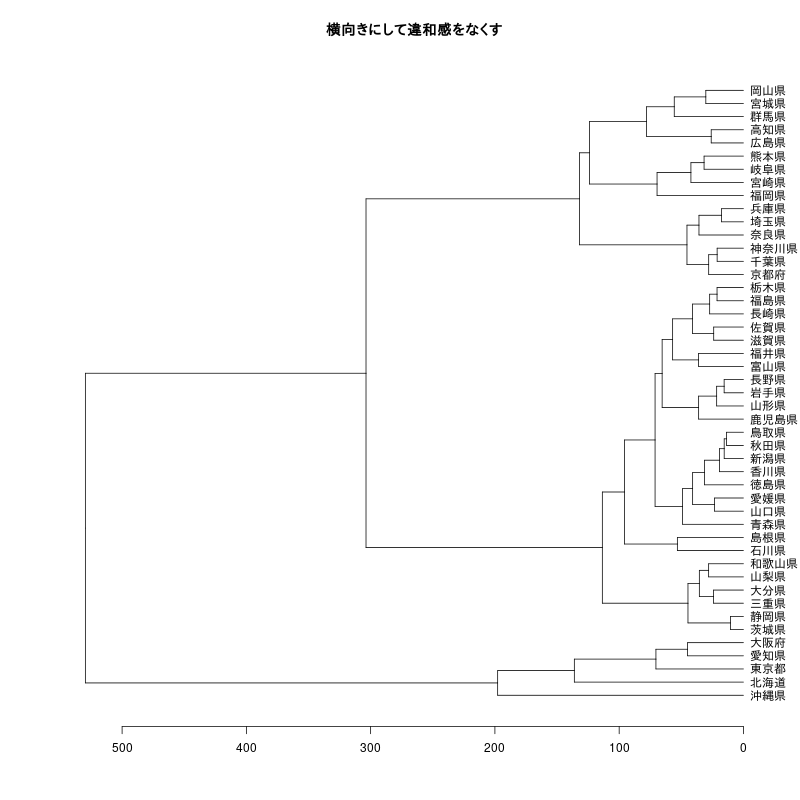
plot(as.dendrogram(h),horiz=T,las=1,main="横向きにして違和感をなくす")
#dev.off()デンドログラム : ラベルを縦書き
#png("Cdendro03.png",width=800,height=600)
par(mar=c(3,5,4,2),cex=1.2)
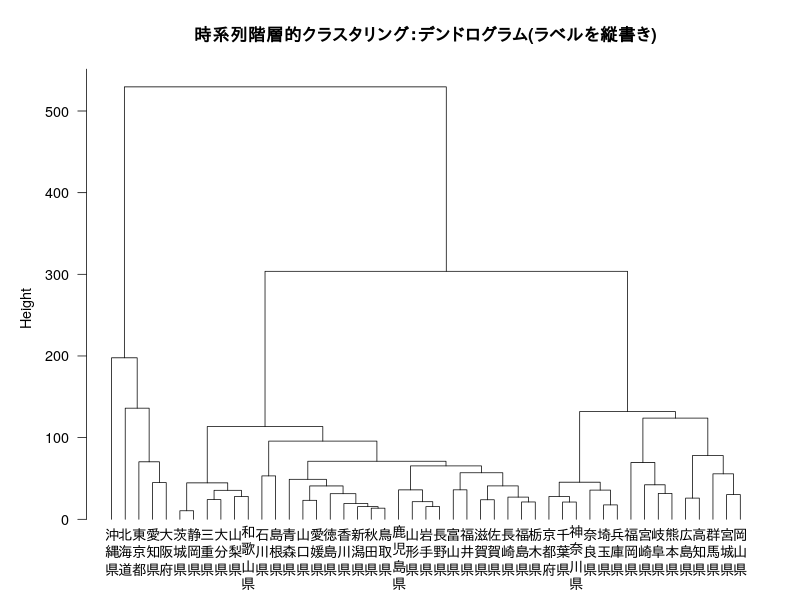
plot(h, labels=F, hang = -1,las=1,main="時系列階層的クラスタリング:デンドログラム(ラベルを縦書き)")
#ラベルを縦書き仕様に
htate<- sapply(strsplit(split="", h$labels), paste, collapse="\n")
# ここがポイント:labels=htate[h$order] でラベルを並び替える。
text(x=1:length(h$labels),y=par("usr")[3]*0.3,labels=htate[h$order],xpd=T,pos=1)
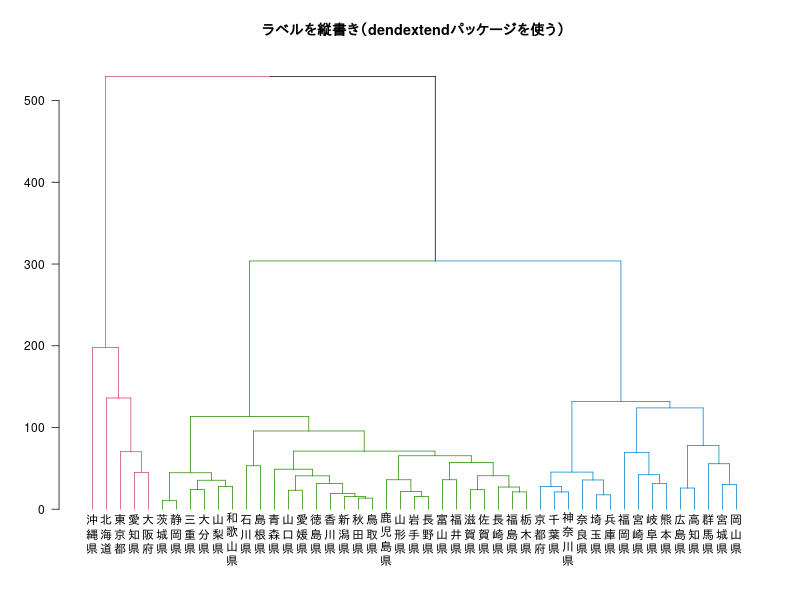
#dev.off()dendextendパッケージを使った場合
library(dendextend)
#
#dend <- as.dendrogram(h)
#せっかくdendextendパッケージを使うので、
dend<- set(as.dendrogram(h),"branches_k_color",k=3)
dend_labels <- labels(dend)
labels(dend) <- rep("",length(labels(dend)))
#png("Cdendro04.png",width=800,height=600)
plot(dend,las=1)
#ラベルを縦書き仕様に
tatelabels<- sapply(strsplit(split="", dend_labels), paste, collapse="\n")
# as.dendrogramとしておくと、labels=tatelabels ラベルの並び替えの必要なし
text(x = 1:length(dend_labels),y=par("usr")[3]*0.3,labels=tatelabels,srt=0,xpd=T,pos=1)
title("ラベルを縦書き(dendextendパッケージを使う)")
#dev.off()