Article Outline
Rでtwinspanの比較(modified TWINSPAN(modif = T) , Standard TWINSPAN(modif = F) )
* twinspanの結果をRでdendrogramで表す方法を思いついたので記事にします。
(トリッキーな方法ですのでご使用は自己責任でお願いします。)
なお、library (twinspan), Jari Oksanen を使うと(従来型twinspanですが)dendrogramを書くことができます。
(参考)
Analysis of community ecology data in R twinspan : David Zelený
Rでtwinspanできるパッケージ
library(twinspanR), David Zelený
- modified TWINSPAN(modif = T) & Standard TWINSPAN(modif = F)
- it can be installed only on Windows platform (since the engine of the library is based on running *.exe file externally)
library (twinspan), Jari Oksanen
- importing the original FORTRAN code into R
twinspanR package のおもしろいところは、RからtwinspanR package付属の「twinspan.exe」を呼び出し、使っているところ。
「twinspan.exe」はwineを使うと、linuxからでも使える。
dendrogram(分割の仕方の違いがよりはっきりとわかります。)
(注意)
「Standard TWINSPAN」の場合は分ける基準はlevelであって、clusterの数ではない。
「modified TWINSPAN」の場合はclusterの数。(これが「modified TWINSPAN」の大きな利点)
dune (Package: vegan)
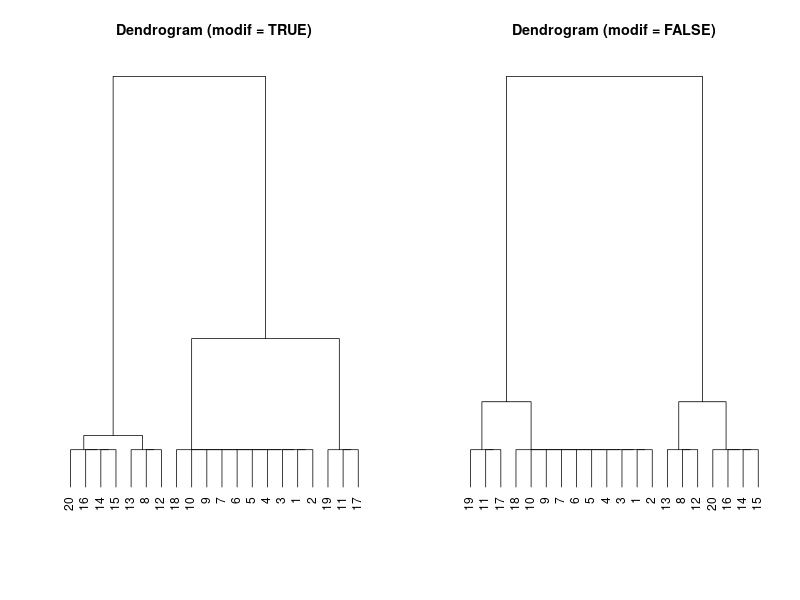
- Standard TWINSPAN はlevel=2で4つに分かれた。modified TWINSPAN は4つに分けた。
mite (Package: vegan)
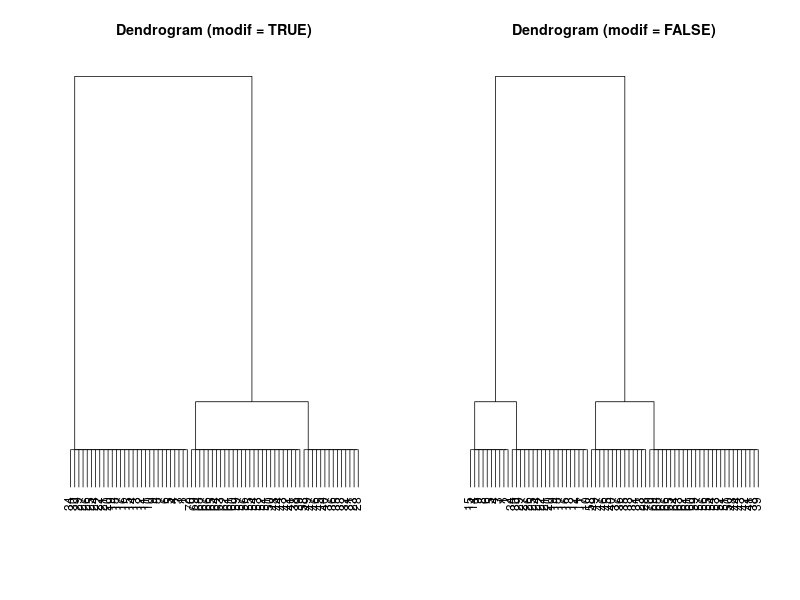
- modified TWINSPAN は3つに分けた、 Standard TWINSPAN はlevel=2で4つに分かれた。
BCI (Package: vegan)
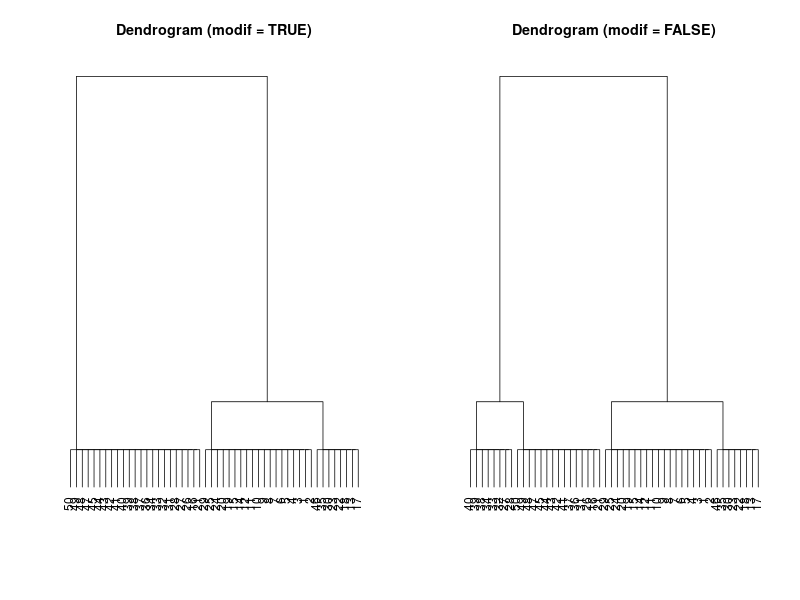
- modified TWINSPAN は3つに分けた、 Standard TWINSPAN はlevel=2で4つに分かれた。
sipoo (Package: vegan)
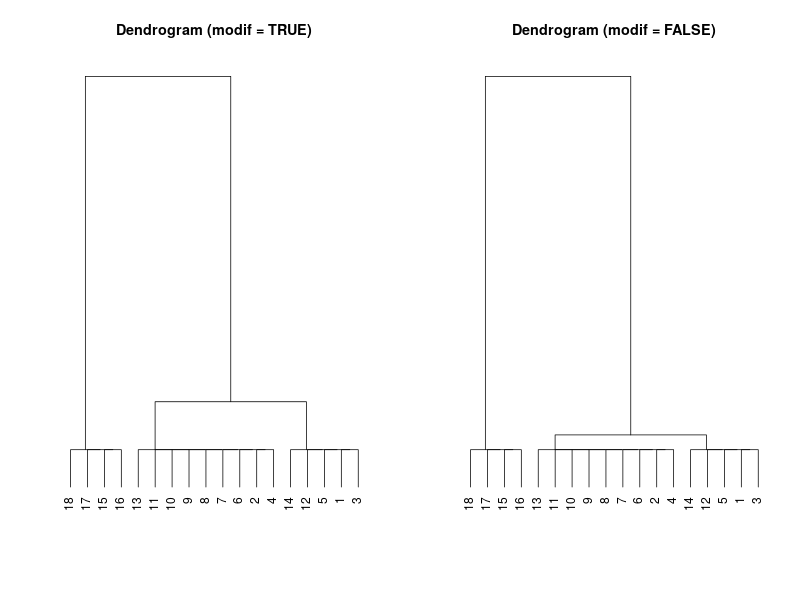
- Standard TWINSPAN はlevel=2で3つに分かれた。modified TWINSPAN は3つに分けた。
pyrifos (Package: vegan)
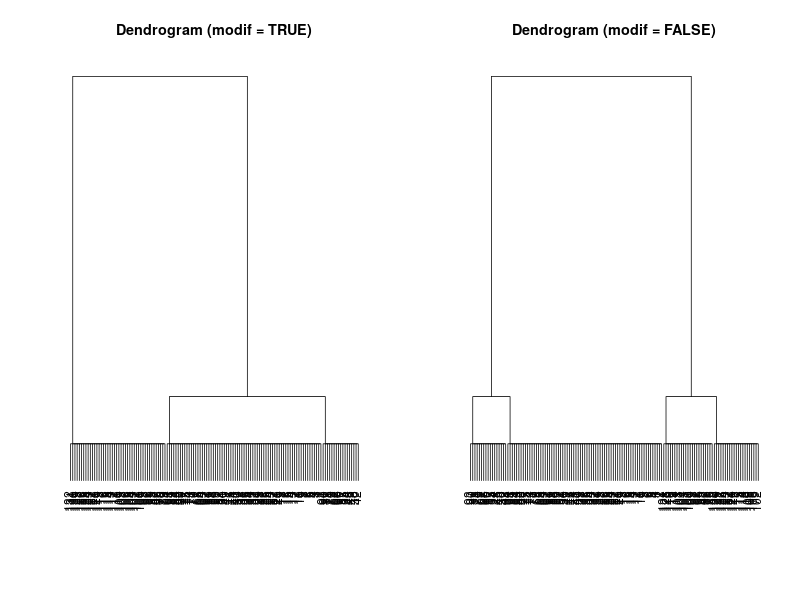
- modified TWINSPAN は3つに分けた、 Standard TWINSPAN はlevel=2で4つに分かれた。
varespec (Package: vegan)
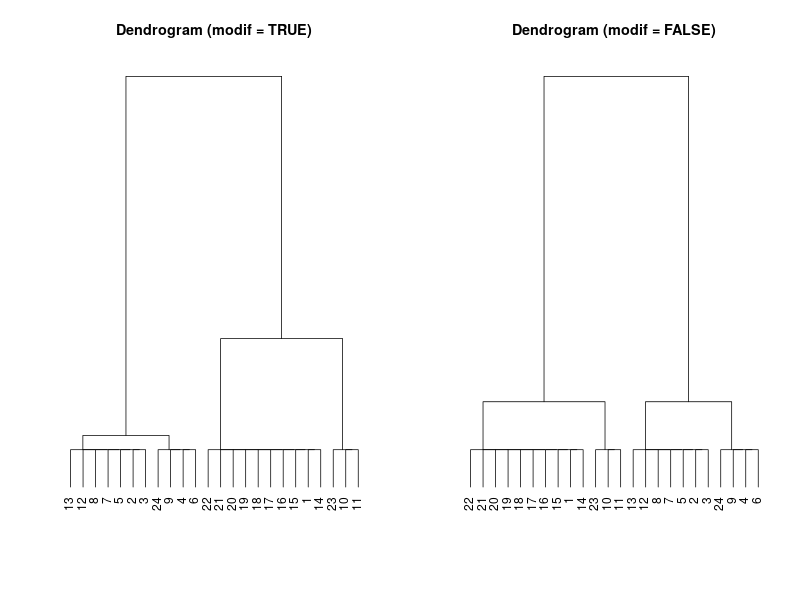
- Standard TWINSPAN はlevel=2で4つに分かれた。modified TWINSPAN は4つに分けた。
danube (Package: twinspanR) <- twinspanR付属のデータセット
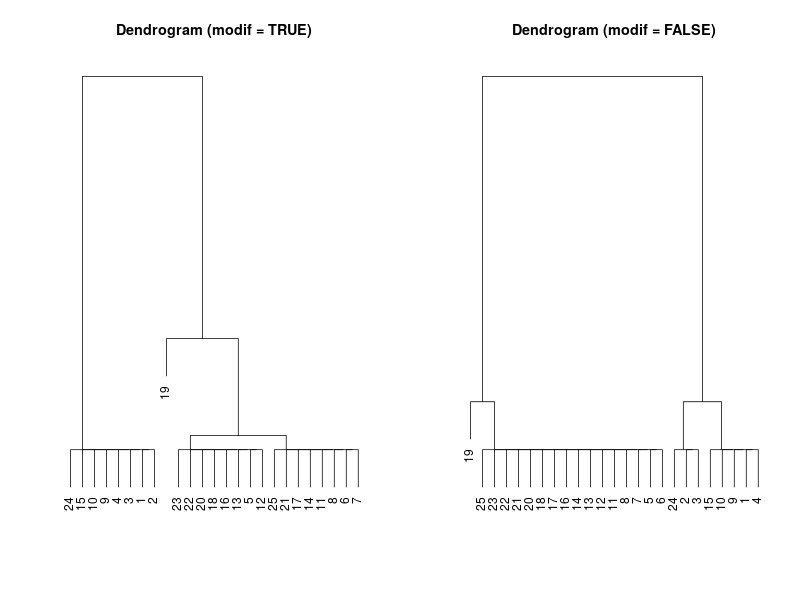
- Standard TWINSPAN はlevel=2で4つに分かれた。modified TWINSPAN は4つに分けた。
Rコード
データセットの読み込み level clusterの数を指定
data(dune)
data<- dune
levels = 2
clusters = 4data(mite)
data<- mite
levels = 2
clusters = 3data(BCI)
data<- BCI
levels = 2
clusters = 3data(sipoo)
data<- sipoo
levels = 2
clusters = 3data(pyrifos)
data<- pyrifos
levels = 2
clusters = 3data(varespec)
data<- varespec
levels = 2
clusters = 4data(danube)
data<- danube$spe
levels = 2
clusters = 4実行
res$classifを参考に手作業でデンドログラムを書くことができます。
*を1 に置き換え、数値に変換する。桁数の大きい箇所の数値(0 or 1)が異なるほど早く分割する。
res <- twinspan(data, modif = TRUE ,clusters =clusters)
tree<- res$classif[,3,drop=F]
rownames(tree)<- as.character(res$classif[,1])
# *を1 に置き換え、数値に変換
tree$class<- as.numeric(gsub("\\*","1",tree$class))
tree<- tree[order(tree$class,decreasing = FALSE),,drop=F]
d<- dist(tree)
# 上下間隔の調整のためlog(d+10)としている。
h<- hclust(log(d+10))
#
res2 <- twinspan(data,modif = FALSE,levels = levels)
tree2<- res2$classif[,3,drop=F]
rownames(tree2)<- as.character(res2$classif[,1])
tree2$class<- as.numeric(gsub("\\*","1",tree2$class))
tree2<- tree2[order(tree2$class,decreasing = FALSE),,drop=F]
d2<- dist(tree2)
h2<- hclust(log(d2+10))
#
#png("tw01_2.png",width=800,height=600)
par (mfrow = c(1,2))
plot(h ,axes=F,ylab = "",sub ="",xlab ="",main="Dendrogram (modif = TRUE)")
plot(h2 ,axes=F,ylab = "",sub ="",xlab ="",main="Dendrogram (modif = FALSE)")
#dev.off()