Article Outline
pgmpyによるベイジアンネットワーク構築から評価、推論まで
# 自分でDAGを考えて指定
my_dag = BayesianNetwork([('A','Y')
, ('B','Y')
, ('C','Y')
, ('A','C')
]
)
# DAG可視化
plt.figure(figsize=(10, 5))
nx.draw_circular( my_dag, with_labels=True, font_family=prop.get_name(), alpha=0.5 )
plt.show()
# DAGをもとにCPD作成
my_dag.fit( df, estimator=BayesianEstimator, prior_type='BDeu' )
# CPD取得
cpd = my_dag.get_cpds()
display(cpd)
print('ノードYの条件付確率表shape', cpd[1].values.shape)
# ノードYの条件付確率表を取得
cpd_status = cpd[1].values
cpd_cols = cpd[1].variables
# ラベルのマスタにノードYのラベルも追加
le_list['Y'] = {0:'negative',1:'positive'}
print(le_list[cpd_cols[0]])
# le_listは{<カラム名>:{<エンコード値>:<ラベル>}}の辞書
# 目的変数の条件付確率が閾値以上の時の条件を取得する
def output_conditional_prob(cpd_status, cpd_cols, prob=0.5, target_status='positive'):
# 条件と条件付確率を格納する辞書
result_dic_1 = {}
# 条件付確率表のShapeのリスト
dim_arr = [[j for j in range(i)] for i in cpd_status.shape]
cnt = 0
# 全条件の組み合わせの確率を取得する
for item in itertools.product(*dim_arr):
# 目的変数がtarget_statusではないとき処理しない
if le_list[cpd_cols[0]][item[0]]!=target_status:
continue
# ある1組の条件と条件付確率を格納する辞書
result_dic = {}
if cpd_status[item]>prob:
print('----', cpd_cols[0], le_list[cpd_cols[0]][item[0]], '----')
# 各変数と各変数の条件をresult_dicに格納
for i, v in enumerate(item[1:]):
print(cpd_cols[i+1], le_list[cpd_cols[i+1]][v])
result_dic[cpd_cols[i+1]] = le_list[cpd_cols[i+1]][v]
print('確率', round(cpd_status[item],3),'\n')
# 条件付確率をresult_dicに格納
result_dic['probability'] = (cpd_status[item])
result_dic_1['Condition_'+str(cnt).zfill(3)] = result_dic
cnt+=1
return result_dic_1
# negative確率が0.8より高い条件
result_dic_0 = output_conditional_prob(cpd_status, cpd_cols, prob=0.8, target_status='negative')
# positive確率が0.8より高い条件
result_dic_1 = output_conditional_prob(cpd_status, cpd_cols, prob=0.5, target_status='positive')
# 推論
infer = VariableElimination(my_dag)
# 推論の実行
results = []
print(len(df))
for num, (index, row) in tqdm(enumerate(df.iterrows())):
new_dict = {k: v for k, v in row.to_dict().items() if k != 'Y'}
result_cpd = infer.query(variables=['Y'], evidence=new_dict, show_progress=False)
results.append(result_cpd.values[-1])
del result_cpd, new_dict
gc.collect()
result_train = pd.DataFrame({'true':df['Y'].to_numpy(), 'pred':results})
display(result_train.head())
# 並列推論の実行
print('All Loop Count', len(df))
# Trainデータ推論(Parallel)
# infer = VariableElimination(my_dag)
infer = BeliefPropagation(my_dag)
pred_values = Parallel(n_jobs=8)(
delayed(infer.query)(
variables=['Y'],
evidence=row.to_dict(),
show_progress=False,
)
for num, (index, row) in tqdm(enumerate(df.drop(columns=['Y']).iterrows()))
)
pred_values = np.array([obj.values[-1] for obj in tqdm(pred_values)])
print(pred_values[:5])
result_train = pd.DataFrame({'true':df['Y'].to_numpy(), 'pred':pred_values})
display(result_train.head())
# Trainデータ評価
y_true = result_train['true'].to_numpy()
y_pred_proba = result_train['pred'].to_numpy()
y_pred = np.round(result_train['pred'].to_numpy())
print(sklearn.metrics.classification_report(y_true, y_pred), '\n')
cm = sklearn.metrics.confusion_matrix(y_true, y_pred)
print('confusion matrix')
print(cm, '\n')
plt.hist(y_pred_proba, bins=20)
plt.title('predict proba hist')
plt.show()
# TestについてTrainには無いカテゴリーはnanに変換しておく
df_test2 = df_test.copy()
df_test2_cols = df_test2.columns
for i, col in tqdm(enumerate(df_test2_cols)):
df_test2.loc[(~df_test2[col].isin(list(df[col].unique()))), col] = np.nan
display(df_test2)
display(df_test2.isnull().sum())
# Test推論(Parallel)
print('All Loop Count', len(df_test2))
# 推論したいノード(変数)以外のノードが欠損だった場合、そのノードも含めたCPDが出力されるイメージ(よって多次元配列が出る)
# infer = VariableElimination(my_dag)
infer = BeliefPropagation(my_dag)
obj_list_test = Parallel(n_jobs=1)(
delayed(infer.query)(
variables=['Y']+row[pd.isna(row)].index.to_list(), # 欠損ノードをvariablesに入れる
evidence=row[~pd.isna(row)].to_dict(), # 欠損ノードはevidenceから抜く
show_progress=False,
)
for num, (index, row) in tqdm(enumerate(df_test2.drop(columns=['Y']).iterrows()))
)
# 任意の軸の‐1番目を取得する関数
def get_zero_index_element(arr, axis, idx=-1):
'''
arr = np.arange(96).reshape(3,4,2,4) # 4dim
print(' dim ', arr.shape)
print('0 dim ', get_zero_index_element(arr, 0, idx=-1).shape)
print('1 dim ', get_zero_index_element(arr, 1, idx=-1).shape)
print('2 dim ', get_zero_index_element(arr, 2, idx=-1).shape)
print('3 dim ', get_zero_index_element(arr, 3, idx=-1).shape)
>> dim (3, 4, 2, 4)
>> 0 dim (4, 2, 4)
>> 1 dim (3, 2, 4)
>> 2 dim (3, 4, 4)
>> 3 dim (3, 4, 2)
'''
slices = [slice(None)] * arr.ndim
slices[axis] = idx
return arr[tuple(slices)]
# 推論したいノード(変数)Y以外のノードが欠損だった場合、欠損じゃないときのノードYの確率のパターンがすべて出力されるので平均を取る
pred_values_test = np.array([np.mean(get_zero_index_element(obj.values, obj.variables.index('Y'), idx=-1)) for obj in tqdm(obj_list_test)])
print(pred_values_test[:5])
# 評価
# stat_test:データの相関の有無を正解値として評価. True=無相関, False=相関
# d_connected:ネットワークのd_connected. True=d結合(相関), False=d分離(無相関)のはずだが、多分逆になっている気がする. True=d分離(無相関), False=d結合(相関)
summary = correlation_score(my_dag, df, test="chi_square", significance_level=0.05, return_summary=True)
summary = summary.rename(columns={'d_connected':'d_separated'})
#summary['d_connected'] = list(map(lambda x: not x, summary['d_connected']))
display(summary)
print('f1_score', np.round(sklearn.metrics.f1_score(summary['stat_test'], summary['d_separated']), 3))
図
notmodel.is_dconnectedだからd_separatedの場合Trueになると思う。correlation_scoreのsummaryのd_connectedはd_separatedの間違いではないか。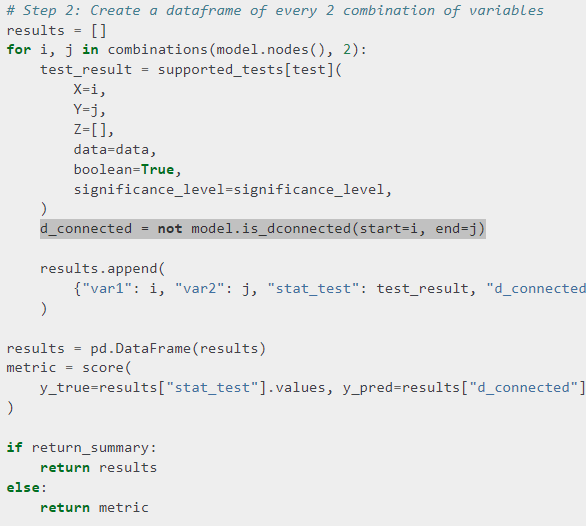
https://pgmpy.org/_modules/pgmpy/metrics/metrics.html
図
boolean = True の場合、検定の p_value が significance_level より大きければ True を返し、そうでなければ False を返す。
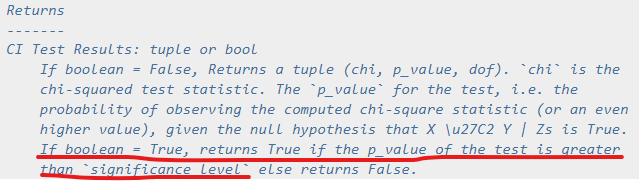
https://pgmpy.org/_modules/pgmpy/estimators/CITests.html